Entwicklungssteuerung durch RHO GTPasen: überraschende Einblicke aus der Welt der Moose
In fast allen eukaryontischen Organismen spielen Enzyme aus der Klasse der RHO GTPasen (Ras Homolog Guanosintriphosphat-Hydrolasen) eine wichtige Rolle in der Steuerung zentraler zellulärer Prozesse, von denen die Entwicklung multizellulärer Gewebe und Organe abhängt. Die Regulation der Polarisierung, gerichteten Expansion, Mobilität und Teilung einzelner Zellen durch RHO GTPasen wurde umfassend untersucht und in vielen Fällen weitgehend aufgeklärt. Dessen ungeachtet gibt es nur wenige direkte Belege für zentrale Funktionen dieser Proteine in der Entwicklung von Tieren oder Pflanzen. Der Nachweis solcher Funktionen beruht auf der phänotypischen Charakterisierung von Mutanten, denen einzelne oder mehrerer RHO GTPasen fehlen („knock-out“ Mutanten). Dieser Ansatz war bisher nur beschränkt zielführend, weil komplexe mehrzellige Organismen typischerweise mehrere eng verwandte RHO GTPasen mit teilweise redundanten Funktionen exprimieren, deren Verlust nur dann phänotypischen Veränderungen hervorruft, wenn alle gleichzeitig fehlen. Zusätzlich scheinen schwere Entwicklungsdefekte die Herstellung bestimmter RHO Mutanten zu erschweren oder zu verunmöglichen.
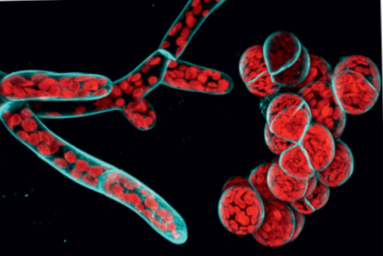
Das Moos Physcomitrella patens ist einer der ganz wenigen multizellulären Organismen, für die direkt gezeigt werden konnte, dass das Fehlen jeglicher RHO Aktivität zu schwerwiegenden Entwicklungsdefekten führt (siehe Abbildung). Ein Team von FAU Zellbiologinnen/-en um Frau PD Dr. Maria Ntefidou hat in Zusammenarbeit mit externen Kolleginnen/-en diese Entwicklungsdefekte systematisch untersucht. Zu diesem Zweck hat das Team sämtliche PpROP (RHO der Pflanzen) Gene in P. patens in allen möglichen Kombinationen ausgeschaltet und die Phänotypen der dadurch generierten „knock-out“ Mutanten, sowie verschiedener komplementierter Varianten dieser Mutanten, umfassend quantitativ charakterisiert.
Die Ergebnisse dieser aufwändigen Studie haben gezeigt, dass die vier untereinander sehr ähnlichen PpROP GTPasen komplett redundante Funktionen haben, und nicht nur wie bereits bekannt Zellexpansion und -verzweigung steuern, sondern auch verschiedene Entwicklungsprozesse (z.B. die Differenzierung bestimmter Zelltypen sowie die Entstehung von Moospflanzen aus den in der Abbildung gezeigten juvenilen filamentösen Strukturen). Interessanterweise hängen verschiedene PpROP-regulierte Prozesse von unterschiedlich hoher Gesamtaktivität aller PpROP GTPasen ab, nicht aber von der Expression spezifischer einzelner Vertreter dieser Proteinfamilie. Zur Validierung dieser überraschenden Erkenntnis hat das Team um Frau PD Dr. Ntefidou überzeugende Hypothesen entwickelt für die evolutionäre Entstehung und Aufrechterhaltung der außergewöhnlich ausgeprägten Aminosäuresequenz-Konservierung und funktionellen Integration innerhalb der PpROP Proteinfamilie.
Originalpublikation
A. Le Bail*, B. Kost*, J. Nüssel, T. I. Lolis, D. Koch, H. Voll, S. Schulmeister, A. Kaier, K. Ljung & M. Ntefidou (2025) Essential developmental processes in Physcomitrium patens require distinct levels of total activity provided by functionally redundant PpROP GTPases. New Phytologist 248, 2865
* equal contribution
https://nph.onlinelibrary.wiley.com/doi/10.1111/nph.70603
Weitere Informationen
PD Dr. Maria Ntefidou
Lehrstuhl Zellbiologie, FAU Department Biologie
09131/85-28222
maria.ntefidou@fau.de
Prof. Dr. Benedikt kost
Lehrstuhl Zellbiologie, FAU Department Biologie
09131/85-28216
benedikt.kost@fau.de